医用画像処理のためのMRI入門
こちらの記事はIPFactory Advent Calendar 2020 5日目の記事です。
非医療従事者が医用画像処理を学ぶにあたって知っておくと良さそうなMRIのことについてまとめました。
筆者は医療についてはド素人です。気を付けて書きましたが、間違っている記述や表現があればご指摘をお願いします。
はじめに
まずは例として以下の論文を見てみましょう。
脳 MR 画像におけるラクナ梗塞の検出法の改良-AdaBoost テンプレートマッチングを用いた偽陽性削除-
この論文の2.実験試料の一部を見てみます。
実験に用いた画像は,岐阜大学附属病院の 1.5T の MR装置(Signa Excite Twin Speed, GE Medical Systems)によって撮影された 132 名の T1 強調画像及び T2 強調画像である.本研究は倫理審査委員会での承認を得た.T1 強調画像の撮影条件は,Spin-Echo 法,TE:8~12 ms,TR:300~500 ms,Slice Width:5 mm,Slice Gap:2 mm であり,T2 強調画像の撮影条件は,Spin-Echo 法,TE:8~12ms,TR:3000~5000 ms,Slice Width:5 mm,Slice Gap:2 mm である.画像サイズは 512×512 pixels,空間分解能は 0.47 mm,年齢は 28~83 歳(平均 63.4 歳),男性 75 名,女性 57 名である.
このように、データセットとして使われた画像についての情報が記述されています。
ある程度流してよい内容も含まれているでしょうが、上記にあるT1強調画像やT2強調画像とはどのようなものでしょうか。
今回はこのように論文に書かれてあるような内容で、前提知識として知っておくべきであろうことを記事にしました。
MRIの概要
「MRI」という言葉や装置のデザインは広く普及しているものの、筆者は装置について何も知らなかったので、簡単にまとめます。
MRIとは、Magnetic Resonance Imaging の略で、磁気共鳴画像(法)と訳されます。
MR装置と表現されたりすることもあります。
MRIは、端的に言えば強い磁場を利用して、体内の水素原子に共鳴反応を起こした際に得られる信号を画像にしているようです。
MRIによって得られる画像は、濃淡のある白黒画像で、三次元的な解析も可能です。
MRIで撮影することは撮像と表現されます(撮影と表現している記事も多く見られたのでそこまで気にかけるものではなさそうですが)。
X線を利用するCTとは、同じ「輪切りにするやつ」でも全然違うものです。
撮像条件
MRIは撮像の条件を変えることで様々な面から人体を検査することができます。
以下に出てくる言葉は、文を読んだり人と話をする中で、当たり前のように出てくる言葉なので、言葉を聞いてすぐに画像の特徴のイメージが湧くことは重要なことだと思います。
基本はT1WI, T2WIで、その2つから応用されたものがいろいろあるようです。
すべての撮像法をまとめているわけではありませんが、様々な情報に触れるなかでよく見るものを取り上げています。
(注: 筆者はまだ頭部MRIにしか触れていないため、脳を中心とした説明になっていきます。)
T1WI(TI強調画像)
T1WIのイメージは、「脳の形がよくわかる画像」です。(こちらのスライドの言葉を借りています)。
解剖学的な構造が見やすく、脳の形態から異常を発見しやすいものとなっています。
英語では、T1-weighted image(s)と表現されます。
T2WI(T2強調画像)
T2WIのイメージは、「病変が白く見える画像」です。(こちらのスライドの言葉を借りています)。
病変が白く映るので、病変の存在を発見しやすいものとなっています。
英語では、T2-weighted image(s)と表現されます。
FLAIR
FLAIRは、脳脊髄液の信号を抑制した(黒く映る)T2WIです。
脳脊髄液の信号を抑制することで、脳の内部の異常が白く強調されるようです。
脳表・脳室付近の病変をFLAIR画像で見ると分かりやすいものとなっています。
ラクナ梗塞をはじめとする無症候性脳梗塞の検査に用いられるようです。
DWI(拡散強調画像)
DWIは、CTでは検出が難しいとされる急性期・超急性期の脳梗塞を発見することができるのが特徴的です。
脳梗塞が新しいものかどうかを知ることができます。
水分子の拡散運動の変化に敏感で、拡散の強さを信号変化として画像にしています。
拡散が低下しているところが高信号として描画され、脳梗塞等が白く見えます。
MRA
MRA(Magnetic Resonance Angiography)は、MRIを用いて血管(動脈系)の血流を撮像するものです。
脳動脈のみが白く映り、血管の形態や血流を把握することができます。
コンピュータによって、血管を3Dで捉えることができるようです。
T2*WI
T2*WI(T2 star 強調画像)は、出血性の病変の検出に向いているとされています。
微小脳出血(Cerebral Microbleeds: CMBs)が明瞭に見えるようです。
過去の脳出血で脳に沈着した鉄分(ヘモジデリン等)が強調されるようで、古い出血の有無の把握に使われています。
SWI(磁化率強調画像)
SWIでは、T2*WIよりも磁化率の変化を鋭敏に捉えることができるようです。
頭蓋内、脳組織においてはデオキシへモグロビン化された静脈血を高精細に描出することもできます(SWI による脳循環の評価:超急性期脳梗塞を中心としてより)。
SWIとT2*WIの比較については、MRI装置による脳内小出血や血管性病変における磁化率強調画像(Susceptibility-Weighted Imaging)とT2*画像(T2* Weighted Image)の比較検討を参照されると良いと思います。
fMRI(機能的MRI)
fMRI(functional MRI)は、MRIを使って脳の活動や機能を調べる方法です。
脳科学辞典 機能的磁気共鳴画像法では、下記のように説明されています。
機能的磁気共鳴画像は、磁気共鳴画像 (magnetic resonance imaging; MRI)を用いて1990年代から脳機能を解明するための研究する技術として使用され、ヒト脳の機能解明に大きく貢献した。当初は脳局所の機能解明法として使用されてきたが、2010年以後は脳局所機能に加え、連絡性を調べる技術として使用されるようになり、その後も全脳の機能・構築・連絡性(コネクトーム)を解明する技術、脳がつかさどる心理状態を解読(デコーディング)する技術、神経難病や精神病態を診断(自動画像診断) する技術 などの開発が急速に進んでいる。まだ臨床診断法として確立した技術ではなく、あくまで研究用として使用されている。
パルスシーケンス
T1WI, T2WI等とは別に、Spin Echo法やGradient Echo法等の撮像方法があります。
同じT1WIでも、Spin Echo法かGradient Echo法かによって画像のコントラストが変わってくるようです。
最も一般的なものはSpin Echo法とされています。
(どこまで理解しておくべきなのかよくわかっていないため)名前だけ挙げておきます。
- Spin Echo(SE)法
- Gradient Echo(GRE)法
- Inversion Recovery(反復回転)法
その他の用語
TRやTEによっても得られる画像のコントラストは変わってきます。
TR
TRは繰り返し時間(Repetition Time)のことです。
TE
TEはエコー時間(Echo Time)のことです。
T(テスラ)
テスラとは、磁場(磁束密度)の強さを表す単位です。
多くの論文では1.5Tか3.0TのMRIが使われている気がします。
1.5Tと3.0Tの違いは、AIC 八重洲クリニック 3テスラMRIと1.5テスラMRIの違いと利用方法にてわかりやすく比較されているので、こちらを参照されると良いと思います。
DICOMファイル
さて、MRIで撮像されたデータは、一般にDICOMというファイル形式で出力されます(他にNIfTI-1やANALYZEというファイル形式もあるようです)。
DICOMは画像でよく見るJPEG等とは少し違うので、見ていきたいと思います。
DICOMの概要
DICOM(ダイコム, Digital Imaging and Communications in Medicine)とは、多くの医療機器で使われる画像のファイル形式や通信プロトコルのことを指します。
MRIだけでなく、CTやPETでも、出力がDICOM形式のファイルとなっています。
拡張子は.dcmです。
DICOMファイルには、撮像(影)された画像だけでなく、そのときの撮影条件(機械の設定だけでなく、患者さんの情報等も)が含まれています。
機器の違いを意識することなく、ユーザは画像や画像に付随する情報を見ることができます。
(少なくともWindowsでは)JPEGやPNG等のようにクリックしたら画像が見れるというわけではありません。
基本的にDICOMファイルを開くためには、DICOMの規格に対応したビューアソフトを利用する必要があります。
DICOMファイルをPythonで扱う
pydicomというPythonのパッケージがあります。
pydicomを利用すれば、PythonでDICOMファイルを扱うことができます。
pydicomの使い方に関しては、kitanote Pydicomの使い方を参照されると良いと思います。
日本放射線技術学会 画像部会がDICOM形式の胸部腫瘤陰影像の標準ディジタル画像データベースを無償で提供してくれていますので、今回はこのデータセットをありがたく利用させていただきます。
例として、メタデータの表示と、画像の表示、1画素あたりのビット数の表示をやってみます。
メタデータの表示
from pydicom import * d = dcmread('./Nodule154images/JPCLN001.dcm') print(d.file_meta)
$ python3 show_metadata.py (0002, 0000) File Meta Information Group Length UL: 230 (0002, 0001) File Meta Information Version OB: b'\x00\x01' (0002, 0002) Media Storage SOP Class UID UI: Secondary Capture Image Storage (0002, 0003) Media Storage SOP Instance UID UI: 1.3.6.1.4.1.9590.100.1.2.281538477311359682300878089351675530135 (0002, 0010) Transfer Syntax UID UI: Explicit VR Little Endian (0002, 0012) Implementation Class UID UI: 1.3.6.1.4.1.9590.100.1.3.100.7.1 (0002, 0013) Implementation Version Name SH: 'MATLAB IPT 7.1' (0002, 0016) Source Application Entity Title AE: 'PAPYRUS 3.0'
画像の表示
import numpy as np import matplotlib.pyplot as plt from pydicom import * d = dcmread('./Nodule154images/JPCLN001.dcm') arr = d.pixel_array plt.imshow(arr, cmap="gray") plt.show()
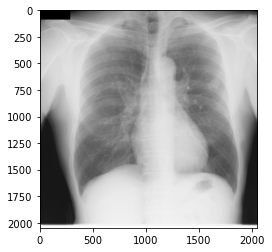
1画素あたりのビット数の表示
from pydicom import * d = dcmread('./Nodule154images/JPCLN001.dcm') print(d[0x0028, 0x0101])
$ python3 show_theNumberofBits.py (0028, 0101) Bits Stored US: 12
DICOMからJPEGへの変換
DICOM形式の画像を機械学習系のネットワークに通す場合、入力画像はJPEGやPNGでの形式がほとんどだと思うので、DICOM形式の画像をなんらかしらの形で変換する必要があります。
以下では、pydicomを使ってDICOMからJPEGに変換することを目標として見ていきます。
変換するには少し工夫が必要でした。
うまくいかなかったソースコードは特に公開しませんが、DICOMファイルを読み込んで、numpy.ndarrayに変換して、.jpgで保存したらオワリみたいな簡単な話ではありませんでした。

このようにほぼ真っ白になってしまいました(データは先ほど画像の表示をしたJPCLN001.dcmです)。
うまくいかない原因は、DICOMとJPEGとで表現の仕方が違うところにあります。
MRIやCTで得られたDICOM形式の画像は、多くの場合16bit等の8bit以上で表現される一方で、JPEGやPNG等は8bitで表現されます
(今回のデータセットは12bitで表現されていました。)。
この違いがあるために、上記の画像のように変換がうまくいかなかったと思われます。
この問題を解決するためには、ウィンドウレベル変換と呼ばれる処理が必要になってきます。
ウィンドウレベル変換
雑な説明となりますが、ウィンドウレベル変換は、ある範囲内の画素値を、目的とする範囲の画素値に変換する処理です。
処理の内容としては、幅(window(, window width))と中心点(level(, window center))を決めて、その範囲の中の画素値を8bitに収まるようにすることをします。
以下のウィンドウレベル変換をしてJPEGにできるコード(アルゴリズム)の大部分は医療情報学研究室 Pyゼミ1.02 DICOMからJPEGなどへの変換から拝借いたしました。
from pydicom import * import cv2 import os # 適切にパスを設定してください。 input_dir = './Nodule154images/' output_dir = './jpg_images/' def main(input_dir, output_dir): list_of_files = os.listdir(input_dir) for dicom_file in list_of_files: d = dcmread(os.path.join(input_dir, dicom_file)) window = d.WindowWidth level = d.WindowCenter arr = d.pixel_array """ JPCLN001.dcmの場合、 window = 4095 level = 2047 だったことより、1画素当たり12bit使われていることがわかる。 """ max_pixelvalue = level + window/2 min_pixelvalue = level - window/2 # 画素値を0~255(8bit)に収まるように変換する。 arr = 255*(arr - min_pixelvalue)/(max_pixelvalue - min_pixelvalue) arr[arr > 255] = 255 arr[arr < 0] = 0 cv2.imwrite(os.path.join(output_dir, dicom_file.replace("dcm", "jpg")), arr) if __name__ == '__main__': main(input_dir, output_dir)
上記のコードを実行すれば、JPEGに変換された画像が得られました。
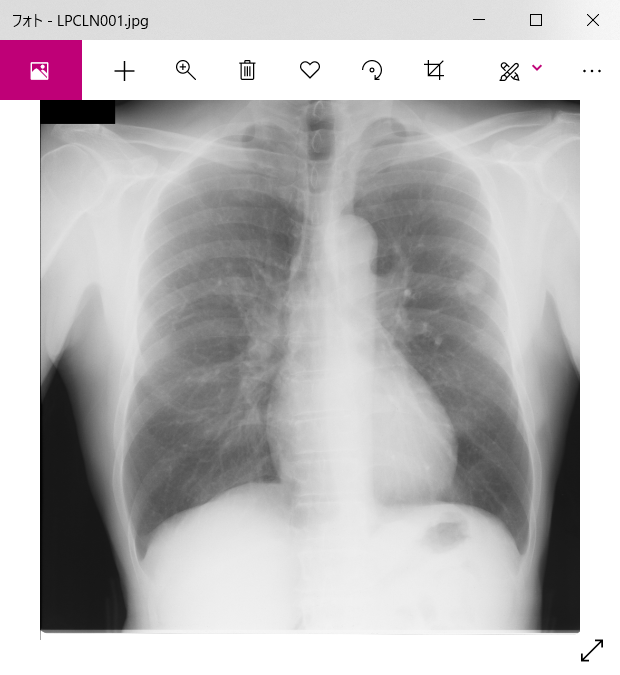
しかし、154枚中、4枚が真っ暗な画像として出力されてしまいました。
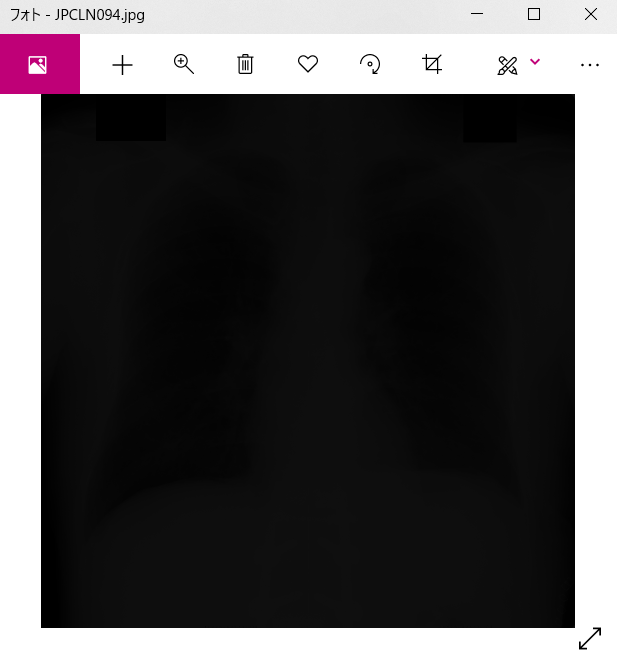
JPCLN094.dcmをmatplotlibで確認してみましたが、他の画像と特に大きな違いはなさそうです。
より最適な書き方があるのかもしれません(原因がよくわからない)。
3次元的に画像を捉えるために
画像を3次元的に扱うための方法の1例として、Voxel-Based Morphometry(VBM) というものが挙げられます。
VBMでは、VoxelというPixelの3次元版(volume + pixelらしい)の情報を利用して、全脳を対象に統計的な解析ができます。
VBMを利用したソフトウェアとして、BADD Softwareや、SPM(Statistical Parametric Mapping)等があります。
まとめと所感
今回は、医用画像を扱う上で、前提知識として知っておくべきものをまとめました。
私は先月勉強を始めたばかりで、分野を俯瞰して見れているわけでは全くないので、まずは抑えるべき情報というものが何なのかまだ把握できておりません(もちろん、知識があることに越したことはなく、特に医療系においては知らないことがあるというのは甘えかもしれませんが)。
いつか勉強しないとなと感じていたので良い機会となりました。頭が少し整理された感じがします。
必要そうなことがあれば追記しておきます。
最後までご覧いただき、ありがとうございました。
https://github.com/futabato/fromDICOMtoJPEG.git
参考
- 脳 MR 画像におけるラクナ梗塞の検出法の改良-AdaBoost テンプレートマッチングを用いた偽陽性削除-
- 一般財団法人 片倉病院 MRI装置と検査
- 頭部MRIを自信を持ってプレゼンする7 Rule
- 核磁気共鳴画像法 (Wikipedia)
- 頭頸部放射線診断学: MRIの撮像法とその信号強度を中心に
- はしぐち脳神経クリニック 脳MRI検査とは
- SWI による脳循環の評価:超急性期脳梗塞を中心として
- MRI装置による脳内小出血や血管性病変における磁化率強調画像(Susceptibility-Weighted Imaging)とT2*画像(T2* Weighted Image)の比較検討
- 脳科学辞典 機能的磁気共鳴画像法
- AIC 八重洲クリニック 3テスラMRIと1.5テスラMRIの違いと利用方法
- pydicom
- 日本放射線技術学会 画像部会 胸部腫瘤陰影像の標準ディジタル画像データベース
- kitanote Pydicomの使い方
- メディカルウェア ウィンドウレベル変換
- 医療情報学研究室 Pyゼミ1.02 DICOMからJPEGなどへの変換
- BADD Software
- SPM(Statistical Parametric Mapping)
余談: IPFactoryについて
IPFactoryとは、情報科学専門学校に存在する学内サークルです。
IPFactoryに在籍するメンバーがそれぞれ自身の専門分野についての勉強やアウトプット活動を行っています。
サークル内有志メンバーでCTFを主催したりしています。
IPFactoryに所属する各メンバーのTwitterやブログ等はこちらのページ を参照してください。
明日6日目はn01e0の「fanotify-rs」です。お楽しみに。